Mensaje Bioquímico XLVI, 2022

Una introducción a la bioinformática: avances en la biología y ciencias de la salud
Red de Apoyo a la Investigación (RAI), Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán Edificio de Radio-Oncología, 2o piso Vasco de Quiroga 15, Belisario Domínguez Sección XVI, Tlalpan C.P. 14080 Ciudad de México.
Tel. +52 (55) 5487 0900 ext. 6327, tobias@cic.unam.mx
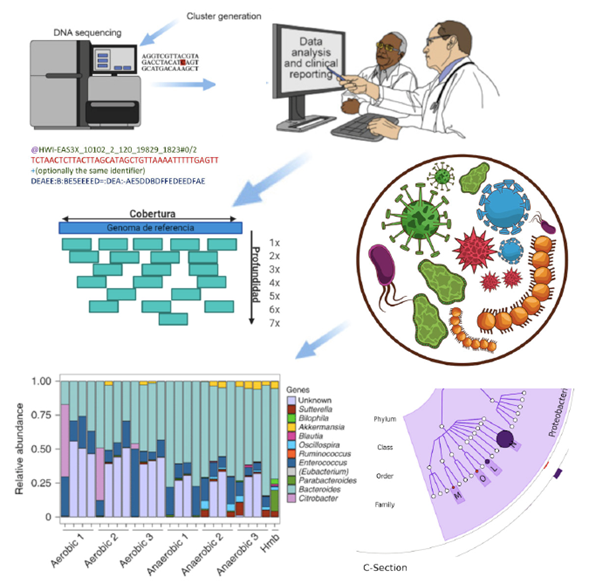
Se presenta una introducción sobre la bioinformática, sus orígenes y su campo de aplicación mostrando sus herramientas computacionales para el análisis de secuencias. Se revisa el estado del arte de las bases de datos o repositorios internacionales de datos biológicos y de las tecnologías de alto rendimiento, con su aplicación en la medicina y ciencias de la salud. Por ejemplo, se aborda el NCBI, PubMed, PDB, Interpro y otras, algunas aplicaciones en la biomedicina y se presenta un protocolo para el análisis de datos de secuenciación masiva (microbiota usando el gen 16S rRNA), detallando el flujo de trabajo, software y requerimientos mínimos de cómputo. Finalmente, se señala el alcance de la bioinformática en la interpretación de los resultados, formulación de hipótesis y en general para el avance de la investigación en la biología y en las ciencias biomédicas.
Técnicas para el estudio del microbioma
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán. Vasco de Quiroga 15, Belisario Domínguez Sección XVI, Tlalpan C.P. 14080 Ciudad de México.
ORCID: 0000-0002-9238-1191
Tel. +52 (55) 5487 0900 ext 2809, palomares.fernanda@gmail.com
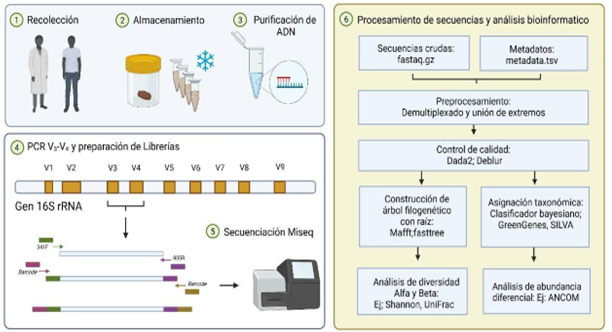
La microbiota intestinal ha sido un importante objeto de estudio en años recientes, dado el amplio número de observaciones que señalan la relación entre las bacterias intestinales y el estado de salud o enfermedad en humanos. La utilización de muestras fecales, es una alternativa metodológica de gran utilidad para estudiar las comunidades microbianas del intestino por su fácil obtención de carácter no invasivo. Sin embargo, el uso de estas muestras comprende distintas consideraciones metodológicas que abarcan la obtención y condiciones de almacenamiento y procesamiento de las muestras como importantes variables a considerar, para poder preservar las características originales de la muestra y su representatividad. Así como la estrategia de secuenciación usando tecnologías de nueva generación y el cómo podemos valernos de recursos bioinformáticos para ayudarnos a conocer la diversidad microbiana de las secuencias derivadas de estas muestras y la presencia de taxones con abundancia diferencial. Por lo tanto, el conocimiento de los retos asociados a la obtención, almacenamiento y procesamiento de muestras fecales y la disponibilidad de técnicas de secuenciación y herramientas bioinformáticas para los análisis de diversidad y taxonómicos, puede ayudarnos a mejorar el diseño experimental al estudiar a la microbiota intestinal.
Metabolómica dirigida para evaluación del perfil de acilcarnitinas en etapas tempranas de la gestación en mujeres que desarrollan diabetes gestacional
Unidad de Vinculación Científica Facultad de Medicina UNAM en el Instituto Nacional de Medicina Genómica (INMEGEN) Periferico Sur 4809, Arenal Tepepan, Tlalpan, 14610 Ciudad de México, CDMX
ORCID: 0000-0001-7600-3046
Tel. +52 (55) 3957 9402, melraz81@gmail.com
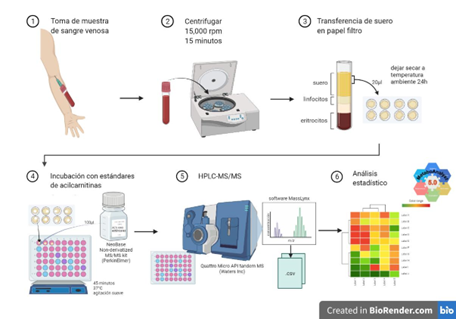
La diabetes gestacional (DG) representa la principal alteración metabólica durante el embarazo, afectando al 17.7% de los embarazos en México. La importancia clínica de esta patología radica en las complicaciones a corto y largo plazo que afectan a la madre y al producto como lo son el desarrollo de diabetes, obesidad, hipertensión y síndrome metabólico. La diabetes gestacional se diagnostica entre la semana 24 a 28 de gestación mediante la realización de una curva de tolerancia a la glucosa. Recientemente, gracias al estudio de la metabolómica se han descubierto diversos biomarcadores que han permitido un mejor entendimiento de las vías metabólicas y metabolitos que derivan en el desarrollo de la diabetes gestacional y que pueden ayudar a desarrollar técnicas y herramientas para el diagnóstico temprano de esta patología en etapas tempranas de la gestación. La metabolómica dirigida ofrece una ventaja para la evaluación y análisis enfocados a preguntas específicas y particulares para evaluar de una manera más eficaz perfiles bioquímicos y moleculares en estados patológicos y no patológicos. Evaluar el perfil de acilcarnitinas en etapas de la gestación en mujeres que desarrollan diabetes gestacional, empleando un enfoque metabolómico dirigido aporta la posibilidad de evaluar e identificar aquellos potenciales biomarcadores clave en el desarrollo de esta patología y su relación con alteraciones en la oxidación de ácidos grasos, aminoácidos, así como la resistencia a la insulina.
Fundamentos de Citometría de Flujo
Instituto Nacional de Medicina Genómica (INMEGEN) Periferico Sur 4809, Arenal Tepepan, Tlalpan, 14610 Ciudad de México, CDMX
Tel. +52 (55) 5350 1900 Ext 1190, lnpatino@inmegen.gob.mx
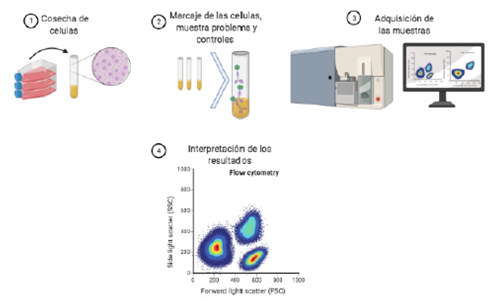
La citometría de flujo es una herramienta biotecnológica que nos permite medir de manera rápida características físicas y químicas de manera individual de cada una de las células suspendidas en una solución. Esta tecnología nos permite evaluar múltiples parámetros de manera simultánea en una muestra, lo que la hace ser una tecnología analítica de gran alcance.En la actualidad es empleada en la clínica, para uso diagnóstico de enfermedades hematológicas e inmunes, así como en la investigación básica, en el desarrollo de experimentos complejos para la comprensión de estudios referentes a la salud. Por lo que es importante tomar en cuenta los requerimientos necesarios para el buen diseño del panel de anticuerpos, el manejo de las muestras y la optimización de los ajustes del citómetro para obtener resultados confiables y precisos.
Anticuerpos antinucleares por inmunofluorescencia indirecta
Centro de Investigación Facultad de Medicina UNAM-UABJO, Facultad de Medicina y Cirugía, Universidad Autónoma “Benito Juárez” de Oaxaca, Oaxaca 68020, México
orcid:0000-0003-4140-4661
Tel. +52 (1 9) 51 1209805, laurapcm@prodigy.net.mx
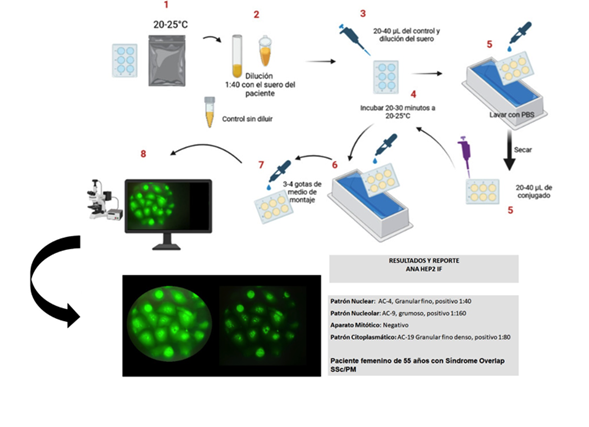
La inmunofluorescencia es una técnica utilizada para estudios de autoinmunidad. La técnica se basa en el reconocimiento de los anticuerpos que reconocen estructuras antigénicas celulares nativas. Esta interacción se pone en evidencia con anticuerpos antiinmunoglobulina humana, ya sea de conejo, cabra o cobayo, que van dirigidos contra las fracciones constantes de las inmunoglobulinas IgG, IgA y/o IgM. Este anticuerpo antiinmunoglobulina humana está conjugado con un fluoróforo como el isotiocianato de fluoresceína. Los resultados del reconocimiento de los antígenos por los autoanticuerpos presentes en el suero, plasma o cualquier otro líquido, se evalúan en un microscopio de fluorescencia.
Cristalografía de macromoléculas
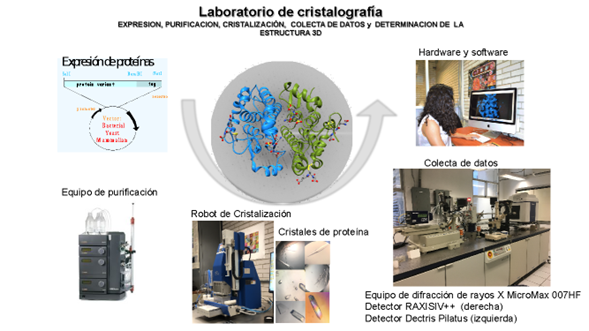
La cristalografía, una disciplina que involucra el uso de técnicas de difracción de rayos X para revelar las estructuras tridimensionales de las proteínas y otras macromoléculas a escala atómica, ha dado lugar a numerosos avances en la comprensión de su funcionamiento en bacterias, virus, animales, plantas y seres humanos. Además, ha guiado el desarrollo de medicamentos y vacunas para combatir diversas enfermedades como alergias, cáncer, SIDA, etc. y mas recientemente el SARS-COV-2. La cristalografía de proteínas es una forma de “microscopía” de muy alta resolución, que permite “ver” las moléculas a una resolución atómica.
En las últimas décadas, los éxitos sin precedentes en este campo han ayudado a muchas disciplinas relacionadas con células y moléculas, en biología y medicina. Las estructuras de enormes complejos como el ribosoma, el centro de reacción fotosintética, las proteínas chaperonas, el complejo de la polimerasa, y los extraordinarios avances en la determinación de estructuras de proteínas integrales de membrana involucradas en el transporte y la conducción de iones, son ejemplos de este avance impresionante.
A la fecha se han logrado avances importantes para superar dos cuellos de botella en este campo: el crecimiento de cristales de alta calidad y la solución del problema de la fase gracias a la robótica, software especializado, equipos caseros mas poderosos, así como el uso de radiación de sincrotrón. En la actualidad, el desafío más crítico al que se enfrenta la cristalografía macromolecular de rayos X es la necesidad de responder preguntas sobre la dinámica de los procesos, con el fin de obtener información sobre los cambios conformacionales que se sabe desempeñan un papel esencial en el control de la función de las proteínas.
Análisis filogenético de Proteínas: Una herramienta básica para la Medicina Evolutiva o Filomedicina
Tel. +52 (55) 5623 2284, hriveros@unam.mx
 Resumen
Resumen
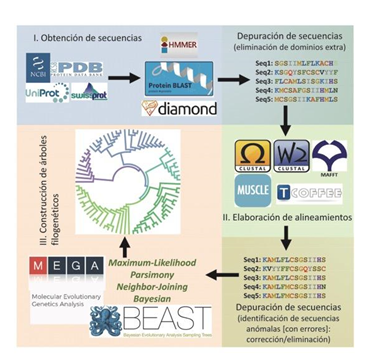
Actualmente, como resultado de la implementación de técnicas de secuenciación masiva, tenemos acceso público a la información de miles de genomas completos y más de 400 millones de secuencias de genes y proteínas. Sin embargo, en menos del 1% de esas proteínas, contamos con información que vaya más allá de un marco de lectura abierto que determine su secuencia primaria. Por esta razón ha sido imperativo implementar métodos de análisis de secuencias y uno de ellos en particular, el análisis filogenético de secuencias ha resultado particularmente exitoso: permitiendo, por ejemplo, discernir entre genes ortólogos y parálogos, estimar tiempos de divergencia, reconstruir proteínas ancestrales, o incluso determinar residuos importantes para la selección natural e identificar mutaciones claves que producen enfermedades o permiten que una proteína adquiera nuevas funciones. Su trascendencia actual va más allá de la Biología Evolutiva, y se ha convertido en una de las herramientas básicas de la Medicina moderna, conformando lo que ahora denominamos como Medicina Evolutiva, Medicina Darwiniana y simplemente Filomedicina. El objetivo de este capítulo es proporcionar algunos lineamientos generales que permitan a los estudiantes que por primera vez realizan un análisis filogenético de proteínas, obtener resultados útiles y confiables.
Inmunotransferencia de geles de proteínas de poliacrilamida tipo Western Blot
Facultad de Química, UNAM. Departamento de Biología, Laboratorio 203, Edificio F. Circuito Escolar s/n, Ciudad Universitaria
ORCID: 0000-0003-2597-9457
Tel. +52 (55) 5622 3899 ext 44449 y 44450, jjgartre@unam.mx
 Resumen
Resumen
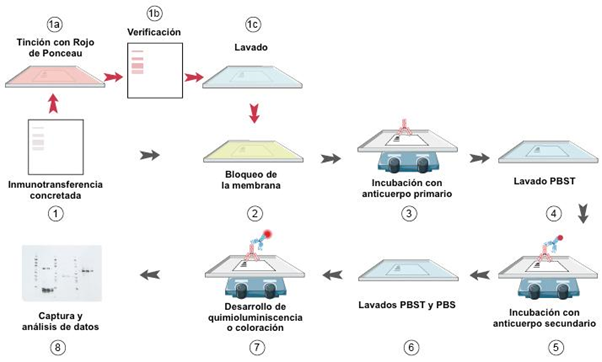
Después de la electroforesis en geles de poliacrilamida o PAGE, la inmunotransferencia o inmunoréplica tipo Western Blot, es una de las técnicas más poderosas en la bioquímica y la biología molecular modernas, dado que permite identificar y en algunos casos cuantificar a las macromoléculas o proteínas separadas previamente en un gel para estudiarlas de manera separada y definir sus propiedades y funciones importantes en los seres vivos. Estas funciones las llevan a cabo en la mayoría de los casos las proteínas en todas las especies, en sus diversas formas como lo son enzimas, receptores, transportadores, bombas primarias y secundarias, proteínas estructurales, etc. Es por ello que posterior a la electroforesis de proteínas o PAGE, la inmunotransferencia tipo Western Blot es una técnica tan poderosa, ya que no sólo permite analizar los niveles de expresión de las proteínas de interés dentro de las células, sino que también permite definir interacciones con otras proteínas, y en general la relevancia de la función de una proteína dentro de cualquier ser vivo. Por todas estas razones, se revisará en este artículo la inmunotransferencia o inmunoréplica tipo Western Blot, posterior a la electroforesis en cuanto a su historia y fundamentos, veremos cómo surgió esta técnica de Western Blot posterior a las técnicas análogas con el ADN o el ARN como lo son el Southern Blot y el Northern Blot, respectivamente, así como los protocolos más comunes y útiles para su uso eficiente en el laboratorio ya sea de investigación, biomédico, farmacéutico, biotecnológico o clínico y diagnóstico.
Consideraciones para la realización de un ensayo enzimático
Departamento de Bioquímica, Facultad de Medicina, UNAM. Laboratorio 8. Circuito Escolar, Ciudad Universitaria, Coyoacán, CDMX. México. CP 04510
ORCID: 0000-0001-6866-4768
Tel. +52 (55) 5623 2169, jrendon@bq.unam.mx
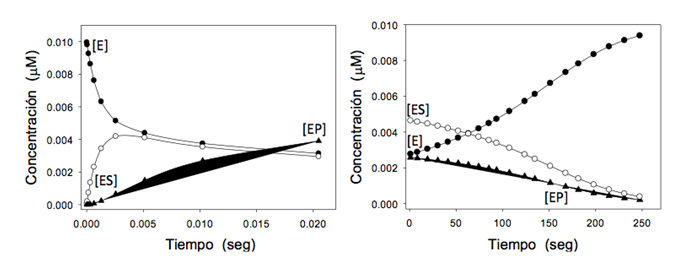 Resumen
Resumen
En el presente trabajo se discuten los diferentes tipos de metodologías disponibles para llevar a cabo un ensayo de actividad enzimática, así como todos aquellos factores que es necesario considerar en el diseño de un protocolo válido. Se enfatiza la importancia de evaluar la velocidad inicial y la razón de su empleo en todo tipo de estudio de cinética enzimática. Adicionalmente, se describen los principios en que se fundamentan los ensayos de actividad directos, indirectos, así como los ensayos acoplados. Finalmente, se presenta el enfoque utilizado para analizar aquellas enzimas que muestran una conducta cinética atípica.
Rreprogramación celular de fibroblastos humanos a células pluripotenciales a través de la transfección de plásmidos episomales
Laboratorio de Tejido Conjuntivo, Centro Nacional de Investigación y Atención de Quemados, Instituto Nacional de Rehabilitación “Luis Guillermo Ibarra Ibarra”. Calzada México-Xochimilco 289, Colonia Arenal de Guadalupe, Tlalpan, México City 14389, México.
ORCID: 0000-0003-2178-9349
Tel. +52 (55) 5999 1000 ext 14701 y 14706, acabrera@inr.gob.mx
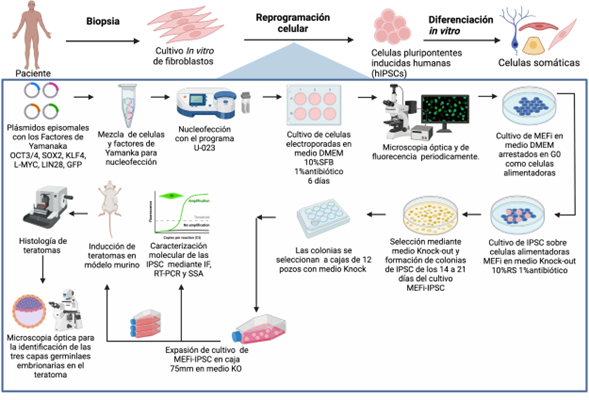 Resumen
Resumen
Desde el descubrimiento de la tecnología de las células pluripotenciales inducidas hace más de 15 años, se abrió un amplio panorama en medicina regenerativa y en el estudio de las células troncales. El proceso de reprogramación resultó ser relativamente sencillo desde una perspectiva técnica, en donde se necesitan una mezcla de 4 factores de trascripción OCT3/4, SOX2, KLF4 y MYC conocidos como OSKM. Esta reprogramación ofrece una oportunidad para generar líneas celulares específicas de pacientes que nos ayuden a prevenir y curar enfermedades. Existen diferentes métodos para generar células troncales pluripotenciales inducidas, se dividen en dos grupos, métodos integrativos y no integrativos. Algunos de estos son más adecuados para el estudio de terapia celular, y otros para hacer más eficiente la reprogramación. En esta ocasión nos basamos en un método no integrativo, el cual consiste en transfectar plásmidos episomales en fibroblastos humanos, para generar células pluripotenciales inducidas.
El uso de la fluorecencia diferencial de barrido en el estudio y caracterización de proteínas
Instituto de fisiología Celular, UNAM, Lab. 201 Ote. Circuito Exterior s/n, Ciudad Universitaria, Coyoacán, CDMX, México. CP. 04510.
Tel. +52 (55) 56 22 56 57, ncabrera@ifc.unam.mx
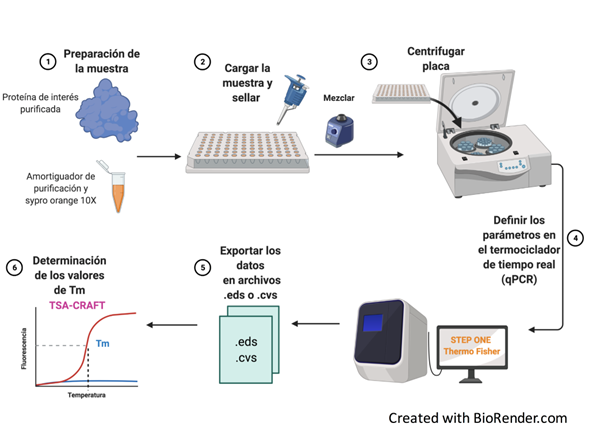 Resumen
Resumen
La fluorimetría diferencial de barrido (Differential scanning fluorimetry, DSF) también conocida como ensayo de desplazamiento térmico (Thermal Shift Assay, TSA) es un método que consiste en la determinación de la temperatura media de desnaturalización de una proteína (Tm) y se determina monitoreando el aumento de fluorescencia del sypro orange, un colorante que tiene afinidad por regiones hidrofóbicas en las proteínas que quedan expuestas a medida que la proteína se va desplegando por el incremento de la temperatura. Mediante un ajuste de la curva obtenida se puede obtener el punto medio de transición que corresponde a la Tm.
Esta técnica tiene numerosas aplicaciones, debido a que se pueden evaluar diferentes variables que ayuden a la estabilización de las proteínas. Por ejemplo, la búsqueda de pHs óptimos, concentración de sales, osmolitos, las mejores condiciones para el almacenamiento de la proteína de estudio y para el mantenimiento de las interacciones proteína-ligando. También, se ha utilizado para analizar el efecto de mutaciones en la estabilidad térmica de las proteínas. En la actualidad, este método es ampliamente usado en ensayos de cristalización y en la búsqueda de moléculas pequeñas con potencial terapéutico. Una de las principales ventajas del TSA es su capacidad para hacer ensayos de alto rendimiento, es decir, evaluar al mismo tiempo múltiples condiciones utilizando placas de 96 o de 380 pozos. Por otro lado, el método está diseñado para llevarse a cabo en un equipo de PCR en tiempo real, lo cual permite obtener resultados en el mismo día a bajo costo. En este artículo se presentan los fundamentos de la técnica, así como los principales aspectos a considerar para realizar ensayos de TS que incluyen el análisis y manejo de los datos para obtener los valores de Tm. Así mismo se presentan algunos ejemplos que describen los alcances de esta técnica.
Microscopio Confocal: consideraciones prácticas para adquirir imágenes de buena calidad
Unidad de Imagenología. Instituto de Fisiología Celular, Universidad Nacional Autónoma de México. Circuito Exterior s/n, Ciudad Universitaria, Coyoacán, CDMX, México. CP. 04510.
ORCID: 0000-0002-5070-938X
Tel. +52 (55) 5622 5610, rrincon@ifc.unam.mx
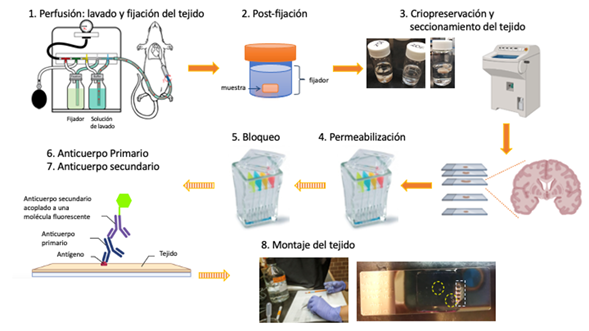 Resumen
Resumen
El sistema CRISPR-Cas9 es una poderosa tecnología que permite editar el genoma eucarionte de manera rápida, precisa y eficiente. Esta herramienta ha revolucionado la manera de modificar el genoma de distintos organismos, desde microorganismos hasta mamíferos. Mediante el uso de CRISPR-Cas no es solo posible introducir mutaciones para estudiar la ausencia de un determinado gene, sino que también nos permite editar el genoma para introducir marcadores fluorescentes o incluso editar el epigenoma. Los protocolos de CRISPR-Cas9 se basan en introducir a las células tanto a la proteína Cas9 como a RNAs guías que dirigen al sistema junto con templados de homología. En este capítulo presentamos protocolos detallados para aplicar esta tecnología de edición genómica en distintos organismos modelo como el nematodo Caenorhabditis elegans, líneas celulares de la mosca de la fruta Drosophila melanogaster, el pez cebra Danio rerio y ovocitos de ratón. Esperamos que este capítulo permita a distintos grupos de investigación aplicar esta poderosa tecnología en sus modelos experimentales

